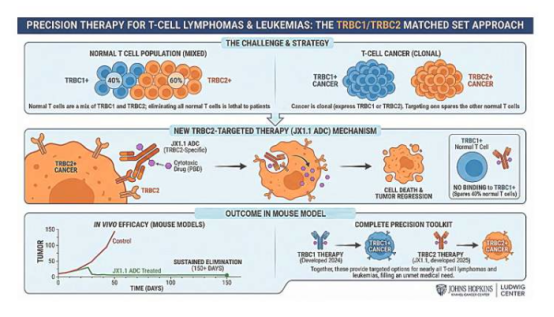
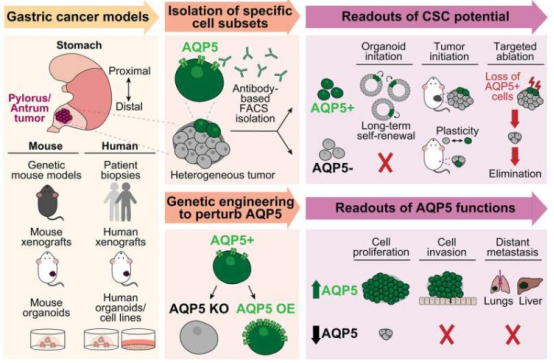
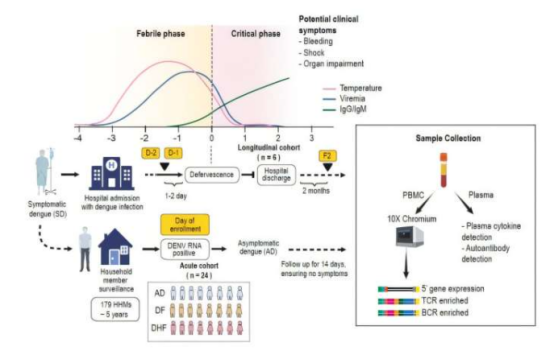
Wissenschaftler konzentrieren sich seit langem auf die Beziehung zwischen Genetik und Krankheiten. Ein Team des Europäischen Laboratoriums für Molekularbiologie (EMBL) hat ein neues Einzelzellsequenzierungstool namens SDR-Seq entwickelt, das Genomvariation und RNA-Expression innerhalb derselben Zelle gleichzeitig erkennen kann. Dieses Tool überwindet die Einschränkungen bestehender Einzelzellsequenzierungstechnologien, verbessert die Erkennungsgenauigkeit und den Durchsatz erheblich und bietet einen neuen Weg zur Erforschung des Zusammenhangs zwischen Variation nicht-kodierender Regionen und Krankheiten.

In einem kürzlich in Nature Methods veröffentlichten Artikel stellte EMBL-Postdoktorand Dominik Lindenhofer fest: „Bestehende Einzelzellmethoden leiden unter begrenztem Durchsatz und unzureichender Empfindlichkeit bei der gleichzeitigen Analyse von DNA und RNA. Unser Einzelzellsequenzierungstool ist nicht durch die Variantenlokalisierung eingeschränkt und kann Einzelzelldaten generieren, die für die Analyse komplexer Proben geeignet sind.“ Über 95 % der krankheitsassoziierten Varianten im Genom befinden sich in nicht-kodierenden Regionen, die mit herkömmlichen Einzelzellsequenzierungstechnologien nur schwer in großem Maßstab analysiert werden können. Das neue Einzelzellsequenzierungstool verwendet ein Öl-Wasser-Emulsionströpfchensystem und ermöglicht die gleichzeitige Analyse des Zusammenhangs zwischen DNA-Variation und Genaktivität in Tausenden von Zellen.
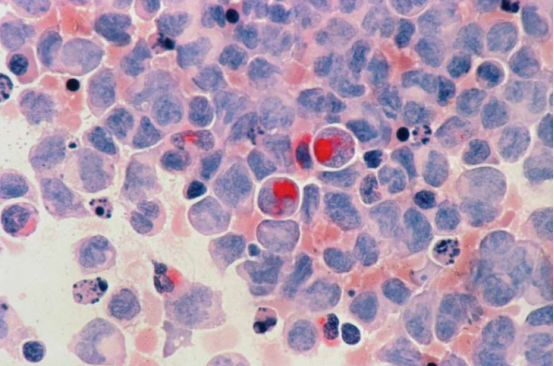
Das Forschungsteam untersuchte B-Zell-Lymphomproben mithilfe von Einzelzellsequenzierungstools und fand heraus, dass die Anzahl genomischer Varianten mit der Bösartigkeit des Tumors zusammenhängt. Lindenhofer ergänzte: „Mithilfe von Einzelzellsequenzierungstools können wir den Einfluss von Varianten auf die Genexpression präzise bestimmen. Beim B-Zell-Lymphom ist die Anhäufung von Varianten mit spezifischen Zellzustandsübergängen verbunden.“ Die Entwicklung dieser Einzelzellsequenzierungstechnologie umfasst eine multidisziplinäre Zusammenarbeit, einschließlich der Optimierung von Zellvorverarbeitungsmethoden und der Entwicklung dedizierter Dekodierungsalgorithmen.
Lars Steinmetz, Gruppenleiter am EMBL, betonte: „Dieses Einzelzellsequenzierungstool stellt eine direkte Verbindung zwischen Varianten und Krankheiten her und eröffnet neue Möglichkeiten zum Verständnis von Krankheitsmechanismen. Durch die detaillierte Analyse von Krankheitsprozessen kann es als Referenz für zukünftige Interventionsstrategien dienen.“ Es wird erwartet, dass diese Technologie bei der Diagnose und dem Screening komplexer Krankheiten eingesetzt wird und die Entwicklung der Präzisionsmedizin fördert.
Weitere Informationen: Dominik Lindenhofer et al., Functional phenotyping of genomic variants using combined multi-omics single-cell DNA-RNA sequencing, Nature Methods (2025). Zeitschrifteninformationen: Nature Methods