Forscher der Universität Regensburg haben durch die Integration experimenteller und rechnergestützter Methoden Fortschritte beim Verständnis der strukturellen und funktionellen Dynamik von RNA-Exosomen erzielt. Die von Teams des Regensburger Zentrums für Biochemie (RCB) und des Regensburger Zentrums für Ultraschnelle Nanomikroskopie (RUN) durchgeführte Forschung wurde in Nature Communications veröffentlicht.
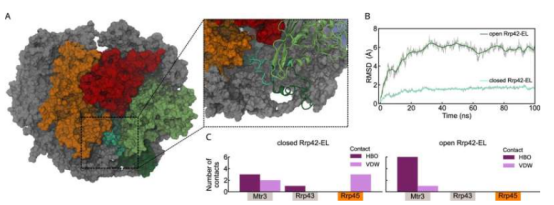
Das Forschungsteam nutzte Kryo-Elektronenmikroskopie, um die statische Struktur von RNA-Exosomen zu bestimmen, und beobachtete darüber hinaus ihre dynamischen Konformationsänderungen durch die Kombination von Kernspinresonanzspektroskopie mit molekulardynamischen Simulationen. RNA-Exosomen sind große Komplexe aus zehn Proteinen, die für den Abbau von RNA in Zellen verantwortlich sind und eine wichtige molekulare Maschinerie zur Aufrechterhaltung der Zellfunktion darstellen.
„Wir konnten die Bewegung bisher unsichtbarer Regionen messen und so die kurzzeitigen Wechselwirkungen zwischen RNA und Exosomen analysieren“, sagte Dr. Jobst Liebau. Dr. Daniela Lazzaretti wies darauf hin, dass die neue Methode es den Forschern ermöglichte, bisher unzugängliche Bereiche des Komplexes zu beobachten. Die Studie enthüllte unterschiedliche Bewegungseigenschaften in verschiedenen Exosomenregionen, darunter langsame Strukturveränderungen, die möglicherweise direkt mit der RNA-Abbaurate korreliert sind.
Professor Remco Sprangers betonte: „Die Kombination verschiedener biophysikalischer Methoden zur Aufklärung der Strukturdynamik wird für die zukünftige Forschung bahnbrechend sein. Wir beginnen gerade erst, die Rolle der Dynamik für die Proteinfunktion zu verstehen.“ Professor Till Rudack erklärte weiter: „Die Kombination von NMR- und molekulardynamischen Simulationen entspricht einem Mikroskop mit extrem hoher räumlicher und zeitlicher Auflösung.“
Diese Forschungsmethode bietet einen neuen Ansatz zum Verständnis der Funktionsmechanismen großer Proteinkomplexe und stellt eine Weiterentwicklung der biophysikalischen Forschungsmethodik dar.
Weitere Informationen: Jobst Liebau et al., 4D-Strukturbiologie – quantitative Dynamik eukaryotischer RNA-Exosomen-Komplexe, Nature Communications (2025). Zeitschrifteninformationen: Nature Communications