Forschende der Cornell University haben einen neuartigen Biosensor namens ProKAS entwickelt. Dieses Werkzeug kann Veränderungen der Aktivität und räumlichen Verteilung von Kinasen in lebenden Zellen mit hoher Präzision erfassen. Dieser Fortschritt in der Technologie zur Kinaseaktivitätsmessung bietet eine neue Methode zur Untersuchung der Zellsignalisierung und der damit verbundenen Arzneimittelentwicklung.
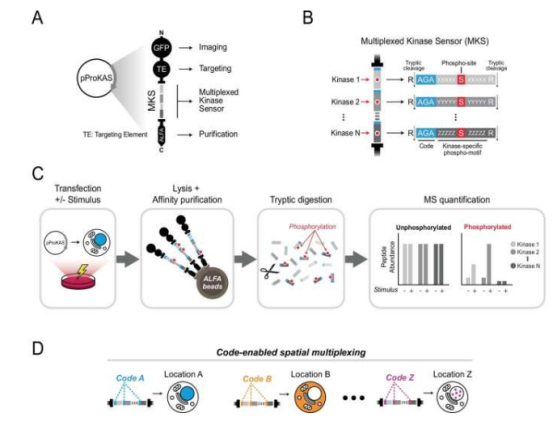
Kinasen sind wichtige Enzyme, die Schlüsselprozesse wie Zellwachstum, Stoffwechsel und Stressreaktionen regulieren. Das Verständnis ihrer genauen Wirkungsweise ist entscheidend für das Verständnis der Zellfunktion und die Entwicklung von Therapien. Bisher fehlten jedoch Werkzeuge, die in der Lage waren, die Aktivität mehrerer Kinasen in lebenden Zellen gleichzeitig und präzise zu lokalisieren und zu analysieren. Die neue Technologie zur Kinaseaktivitätsmessung zielt darauf ab, diese Herausforderung zu bewältigen.
Die Forschung wurde vom Labor von Marcus Smolka, Professor für Molekularbiologie und Genetik an der Cornell University, geleitet, und die Ergebnisse wurden in der Fachzeitschrift *Nature Communications* veröffentlicht. Professor Smolka erklärte: „Angesichts der Herausforderungen bestehender Technologien mussten wir die Messung der Kinaseaktivität und die räumliche Auflösung grundlegend überdenken.“ Er führte weiter aus: „In diesem Fall nutzen wir Massenspektrometrie zur Aktivitätsmessung. Dies ist ein anderer Ansatz zur Verfolgung intrazellulärer Kinaseaktivität als die derzeit verwendeten mikroskopischen Verfahren.“ Kernstück des ProKAS-Systems sind gentechnisch veränderte Peptidketten, die die natürlichen Substrate von Kinasen nachahmen. Jede Peptidkette trägt einen einzigartigen Aminosäure-„Barcode“, der ihren Ort innerhalb der Zelle markiert. Wenn Kinasen auf diese Peptide einwirken, können Forscher mithilfe der Massenspektrometrie gleichzeitig das Aktivitätssignal und den entsprechenden Orts-Barcode erfassen und so die räumlich-zeitliche Verteilung der Kinaseaktivität abbilden.
In Validierungsstudien nutzte das Team diese Technologie, um die dynamischen Reaktionen verschiedener Kinasen auf DNA-schädigende Medikamente zu untersuchen. Sie beobachteten detaillierte Veränderungen der Aktivität wichtiger Kinasen wie ATR und ATM in verschiedenen Bereichen des Zellkerns. Will Comstock, Erstautor der Studie, merkte an: „Wir verarbeiten derzeit Hunderte von Proben, und unser Ziel ist es, zukünftig Hunderte oder sogar Tausende von Proben analysieren zu können.“ Professor Smolka ist überzeugt, dass diese Technologie zur Detektion von Kinaseaktivität ein breites Anwendungspotenzial besitzt. Er erklärte: „Dies ist eine Hochdurchsatzmethode für Pharmaunternehmen, die die Wirkung von experimentellen Medikamenten untersuchen möchten. Forscher können damit schnell Screenings durchführen und die Wirkmechanismen von Medikamenten bestimmen. Ich halte dies für äußerst wertvoll.“ Das Forschungsteam plant, diese Technologie zukünftig mit computergestützten Werkzeugen und anderen Methoden zu kombinieren, um das Verständnis des Kinase-regulierten Zellverhaltens zu vertiefen.
Weitere Informationen: William J. Comstock et al., „Proteomics Sensors for Quantitative Multiplexing and Spatial Monitoring of Kinase Signaling“, *Nature Communications* (2025). Zeitschrifteninformationen: *Nature Communications*.