Das Team von Professor Dominik Niopek am Institut für Pharmazie und Molekulare Biotechnologie (IPMB) der Universität Heidelberg hat ein neues KI-Modell namens ProDomino entwickelt, das den Aufbau von Proteinuntereinheiten vorhersagt und so das Design künstlicher Proteine mit spezifischen Funktionen ermöglicht. Diese in Nature Methods veröffentlichte Forschung liefert neue Forschungsinstrumente für Biotechnologie und Medizin.
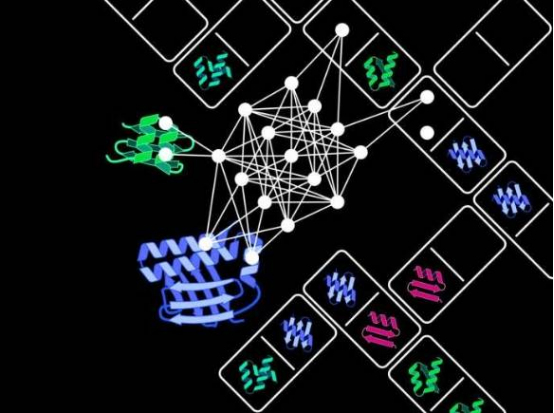
Proteine sind die molekularen Maschinen der Zelle. Sie bestehen aus mehreren Domänen (Untereinheiten), die für Schlüsselfunktionen wie Energiestoffwechsel und Gensynthese verantwortlich sind. Inspiriert von der Proteinevolution in der Natur nutzte Professor Niopeks Team KI-Modelle zur Simulation der Domänenrekombination und entwickelte das Open-Source-Tool ProDomino. Dieses Tool analysiert einen Datensatz von über 100.000 Proteinen, um optimale Domänenkombinationen für die Konstruktion neuartiger Proteine mit steuerbaren Funktionen vorherzusagen.
Das Forschungsteam hat ProDomino bereits in der Praxis eingesetzt, beispielsweise durch die Kombination chemisch sensitiver Domänen mit dem CRISPR-Cas-System zur Entwicklung eines regulierten Genomeditierungswerkzeugs. „Diese Hybridproteine lassen sich präzise ein- und ausschalten, was die Sicherheit der Genomeditierung erhöht“, sagte Projektleiter Dr. Jan Mathony. Das Modell wurde an verschiedenen Proteinen validiert und steht nun Forschern weltweit zur Verfügung.
Professor Niopek betonte: „ProDomino vereinfacht nicht nur die Herstellung künstlicher Proteine, sondern verbessert auch die Designgenauigkeit und eröffnet so neue Wege für biomedizinische Anwendungen.“ In Zukunft könnte diese Technologie die Entwicklung maßgeschneiderter Behandlungen fördern.
Weitere Informationen: Benedict Wolf et al., Rational engineering of allosteric protein switches by computational prediction of domain insertion sites, Nature Methods (2025). Zeitschrifteninformationen: Nature Methods